Как благодаря WebAssembly получилось ускорить приложение в 20 раз
В этой статье мы рассмотрим реальный случай, когда команде удалось ускорить своё браузерное приложение, заменив медленные вычисления JavaScript на предкомпилированный WebAssembly.
Что такое WebAssembly?
Если коротко, WebAssembly (Wasm) — это новый формат инструкций, который можно исполнять в браузере так же, как JavaScript.
Огромное значение имеет то, что вы можете получить код на WebAssembly путём компиляции исходников на C/C++, Rust, Go и многих других языках. В Wasm используется статическая типизация и плоская модель памяти, а сам код хранится в компактном бинарном формате. Из-за этого код выполняется достаточно быстро, почти так же быстро, как если бы вы запустили приложение из командной строки.
Возможность использования уже готовых и известных инструментов и библиотек для веб-приложений и значительный прирост производительности по сравнению с JS делают Wasm очень привлекательным для веб-разработки.
В каких областях используют Wasm?
WebAssembly применяют для разработки приложений из самых разных областей. Он используется как в играх (например Doom 3), так и для портирования известных десктопных приложений (например Autocad или Figma). Даже если вам не нужно веб-приложение, Wasm можно использовать как эффективный и гибкий инструмент для serverless вычислений.
В этой статье будет рассмотрен опыт использования Wasm для ускорения веб-инструмента анализа данных. Мы возьмём уже существующий инструмент на C, скомпилируем его в WebAssembly и используем для замены медленных участков JS.
Что за приложение для анализа данных?
Приложение, о котором пойдёт речь, — fastq.bio. Это интерактивный браузерный инструмент, который показывает учёным, насколько качественные данные секвенирования ДНК были ими получены. Секвенирование — это процесс, при котором считываются «буквы» (точнее, нуклеотиды) образца ДНК.
Мы не будем вдаваться в подробности вычислений, но в общих чертах эта инфографика даёт учёным наглядное представление о том, насколько хорошо прошло секвенирование и есть ли проблемы с качеством данных.
Конечно, есть множество десктопных инструментов, которые позволяют сгенерировать подобные отчёты. Цель fastq.bio — дать интерактивное представление качества данных прямо в бразуере. Это особенно полезно для учёных, которые не очень хорошо владеют командной строкой.
Входные данные для приложения поставляются в виде обычного текстового файла. Он генерируется инструментами, используемыми для секвенирования, и содержит список последовательностей ДНК и оценку качества для каждого нуклеотида в последовательности. Формат файла называется .FASTQ, отсюда и название приложения.
Как это было реализовано с помощью JS?
В изначальной версии fastq.bio пользователь начинал с выбора FASTQ-файла на своём компьютере. Используя объект File, приложение считывало небольшой фрагмент данных начиная со случайной позиции (с помощью API FileReader). Обрабатывая этот фрагмент данных, JavaScript выполнял несложные строковые операции и считал нужные показатели. Один из таких показателей — количество нуклеотидов A, C, G и T на каждой из позиций фрагмента ДНК.
Как только все необходимые показатели посчитаны, они отображаются в инфографике с помощью Plotly.js, а приложение переходит к следующему фрагменту файла. Файл разделяется на фрагменты для улучшения UX: обработка файла целиком занимает очень значительное время, так как FASTQ-файлы обычно весят сотни гигабайт. По опыту можно сказать, что оптимальный размер фрагмента — от 0,5 до 1 Мбайт — при таком объёме приложение будет обновлять инфографику достаточно плавно и у пользователя не будет возникать дискомфорта. Однако при выборе размера стоит учитывать, насколько сложные вычисления производит именно ваше приложение.
Архитектура приложения на JavaScript была достаточно прямолинейна:
А как получится на WebAssembly?
Чтобы понять, можно ли использовать Wasm для оптимизации этого приложения, команда стала искать готовые решения для построения QC-инфографики (quality control, контроль качества) FASTQ-файлов. Искать нужно было инструменты, написанные на C, C++ или Rust, чтобы их можно было лёгким движением руки портировать на WebAssembly. В то же время инструмент должен был быть достаточно надёжным и уже проверенным научным сообществом.
После недолгих поисков было решено остановиться на seqtk. Это приложение достаточно часто используют, у него открытый исходный код, и оно написано на C — подходит по всем параметрам.
Прежде чем компилировать его в WebAssembly, посмотрим, как мы бы компилировали seqtk для использования на десктопе. Согласно Makefile, вот параметры gcc, которые нам нужны:
Чтобы скомпилировать seqtk в WebAssembly, нам необходим Emscripten. Он предоставляет замены существующим инструментам сборки, чтобы работать с WebAssmebly было проще. Если Emscripten у вас не установлен, вы можете воспользоваться образом Docker.
Вы можете также собрать его самостоятельно, но это обычно занимает значительное время.
Внутри контейнера мы можем использовать emcc как прямую замену gcc:
Как вы можете видеть, изменения по сравнению с компиляцией в бинарный файл минимальны:
- Вместо вывода в бинарный файл мы просим Emscripten сгенерировать файлы .wasm и .js. Последний отвечает за запуск модуля WebAssembly.
- Для поддержки библиотеки zlib необходимо использовать флаг
USE_ZLIB. Эту библиотеку используют настолько часто, что она уже портирована на WebAssembly, и Emscripten просто включит её в наш проект. - Мы включаем виртуальную файловую систему Emscrippten. Это POSIX-подобная ФС (исходный код), правда работает она в оперативной памяти внутри браузера и очищается, когда вы обновляете страницу (кроме случаев, когда вы сохраняете состояние в IndexedDB, но это уже тема для отдельной статьи).
Зачем нам виртуальная файловая система? Для ответа на этот вопрос давайте сравним, как мы запускаем seqtk из коммандной строки и как мы запускаем скомпилированный модуль WebAssembly с помощью JS:
Доступ к виртуальной файловой системе очень полезен потому что тогда нам не придётся переписывать seqtk под использование строкового, а не файлового ввода. Мы можем просто отобразить фрагмент данных как файл data.fastq в виртуальной ФС и вызвать main() seqtk на нём.
С использованием seqtk, скомпилированного в WebAssembly, архитектура преобразилась:
Как видно из диаграммы, вместо запуска вычислений в основном потоке браузера мы используем WebWorkers. Это позволяет выполнять вычисления в фоновом потоке, не слишком ухудшая отзывчивость бразузера. Контроллер WebWorker’а запускает Worker и управляет его взаимодействием с основным потоком.
Мы можем попросить Worker’а запустить команду seqtk на файле, который мы только что примонтировали. Когда seqtk закончит выполнение, Worker отошлёт результаты назад в виде Promise. Как только основной поток получит сообщение, он сразу использует его, чтобы обновить инфографику. Как и в реализации через JavaScript, мы обрабатываем файл фрагментами и обновляем картинку на каждой итерации.
Оно того стоит? На WebAssembly получилось быстрее?
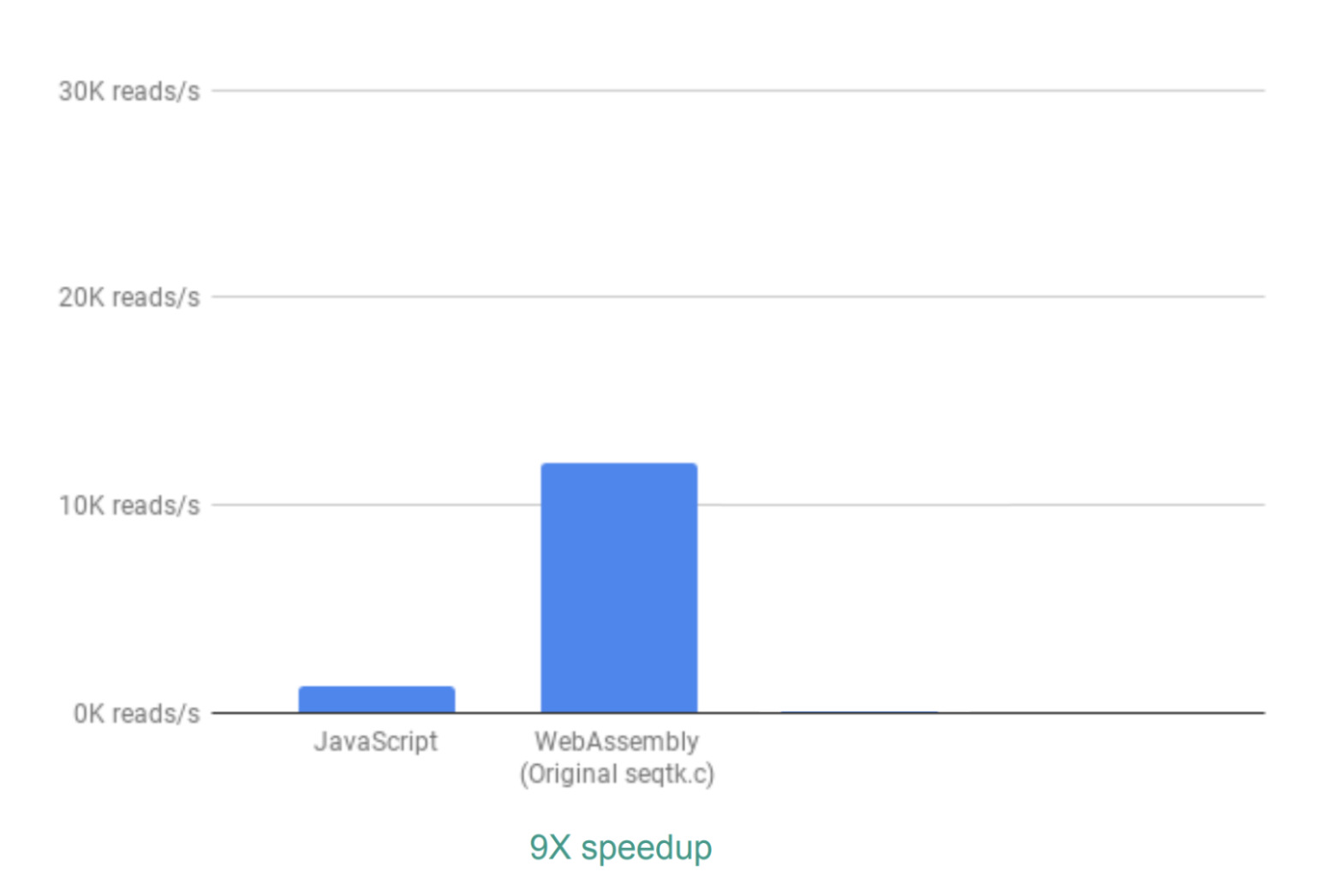
Да. Чтобы понять, принесла ли замена JS на Wasm свои плоды, команда сравнила реализации по параметру «количество операций чтения в секунду». Время, которое уходит на генерацию интерактивных графиков, не учитывается, потому что в обоих случаях для этой части используется JS.
Используя решение «из коробки» команда получила прирост производительности в 9 раз:
Это уже довольно неплохой результат. Особенно учитывая, как просто его было достичь (а его очень просто достичь, как только вы поймёте, как работает WebAssembly).
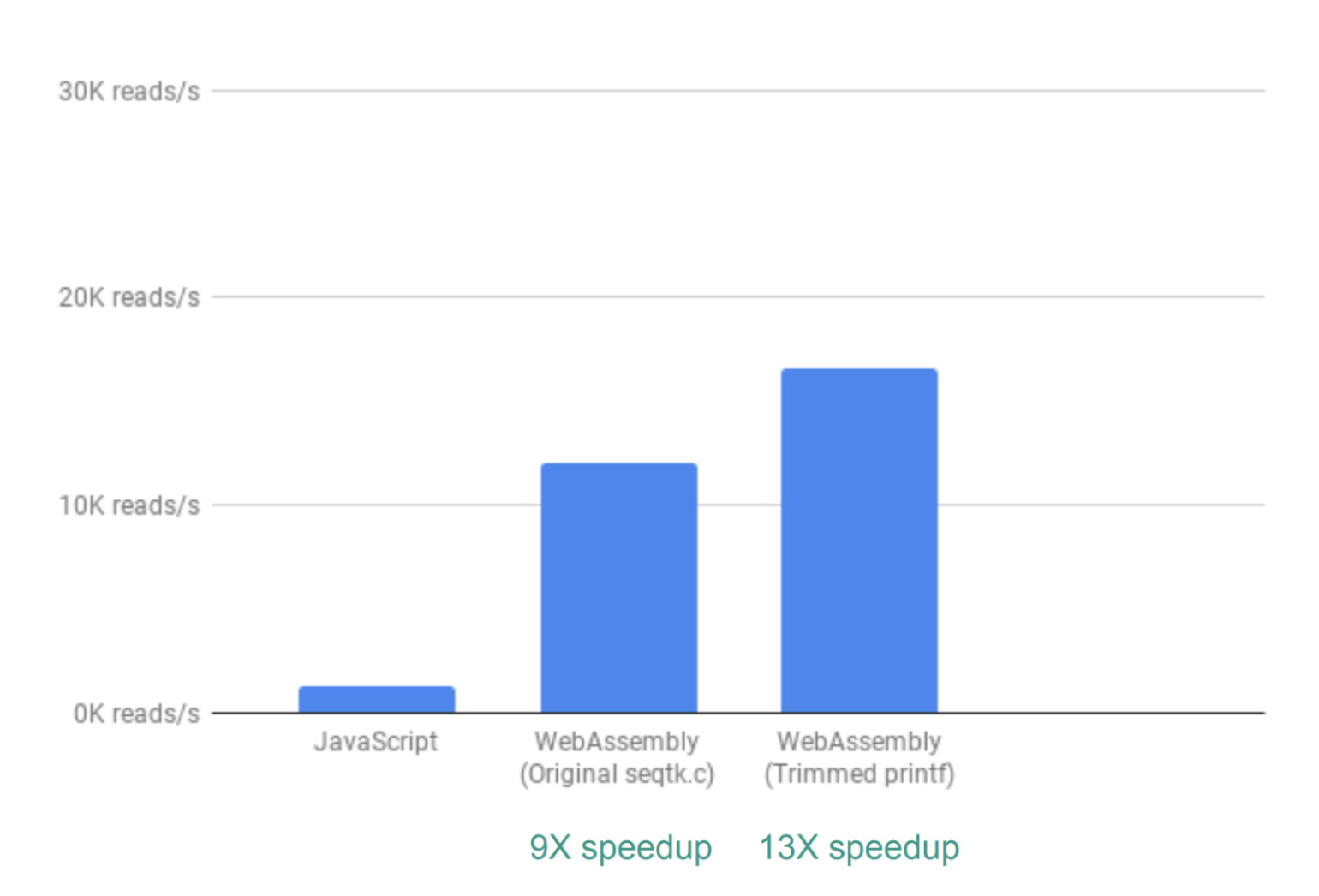
После этого разработчики заметили, что seqtk выводит очень много полезных результатов QC-анализа, но их большая часть не используется в инфографике приложения. После удаления вывода этих параметров результат версии на JS удалось превзойти в 13 раз:
Но на этом история не закончилась. На данном этапе fastq.bio получал результаты анализа вызывая две различные функции C, каждая из которых вычисляла свой набор характеристик. К сожалению, это означало, что каждый фрагмент файла считывается по два раза, хотя это совсем не обязательно.
Решение нашлось довольно быстро — совместить две эти функции в одну. В результате функция получилась ужасна, но знания C особо не потребовалось. Результаты работы функции пришлось сначала совмещать, а потом дополнительно обрабатывать, чтобы разделить назад. Однако это того стоило и позволило увеличить производительность в 20 раз по сравнению с результатом JS.
И что, подобного выигрыша получится достичь всегда?
Конечно нет. Двадцатикратного прироста производительности от простой замены JS на Wasm ожидать не стоит. Вы можете получить ускорение в 2 раза или на 20 %… На самом деле, ваше приложение может наоборот замедлиться, если вы будете загружать большие файлы в память или если ваш код будет требовать большого числа взаимодействий между WebAssembly и JavaScript.
Заключение
Мы увидели, что замена медленного JS-кода на вызовы предкомпилированного Wasm может привести к значительному улучшению производительности. Когда код, необходимый для вычислений, уже написан на C, из этого можно извлечь огромную выгоду. Однако, как уже было сказано выше, WebAssembly — не всегда правильный инструмент для ваших целей. Используйте его с умом.
Дополнительные ресурсы для изучения
- «Level Up With WebAssembly» — практическое руководство по созданию приложений на WebAssembly.
- Aioli (GitHub) — фреймворк для создания быстрых браузерных инструментов для геномики.
- Исходный код fastq.bio (GitHub) — интерактивный онлайн-инструмент для контроля качества результатов секвенирования ДНК.
- «An Abridged Cartoon Introduction To WebAssembly» — краткое введение в WebAssembly с милыми картинками.
12К открытий12К показов